| Phänotypen und genetische Marker der Makrolid-Resistenz von Viridans-Streptokokken, isoliert aus Blutkulturen |
|
C. Jebelean,
L. BindeI, R. Watschinger, M. Haditsch, A. Bocksrucker, H. Mittermayer |
|
Kurzfassung Methoden Ergebnisse Schlussfolgerung |
Einleitung Makrolid-Antibiotika, die als Muttersubstanz Erythromycin haben, sind eine wichtige Alternative bei der Therapie und Prophylaxe von Streptokokken-Infektionen für Patienten mit einer Allergie gegen Penicillin und andere Betalaktam-Antibiotika. Bei Viridans-Streptokokken ist die Prophylaxe der infektiösen Endokarditis ein klassisches Beispiel dafür. Die Erythromycin-Resistenz bei den Streptokokken kann nach einer ribosomalen Änderung durch eine Methylase, die von einem erm-Gen gesteuert wird, oder durch ein Effluxsystem, das von einem mef-Gen gesteuert wird, entstehen. Das erm-Gen kodiert die Erythromycin-Resistenz und eine konstitutive oder induzierbare Resistenz gegen Clindamycin und Streptogramine B (MLSB-Resistenz), wohingegen das mef-Gen nur eine Erythromycin-Resistenz kodiert (M-Resistenz) [1, 2, 3, 4]. Das Efflux-System betrifft nur 14- und 15-gliedrige Makrolide, nicht aber 16-gliedrige wie Josamycin. Beim Vorhandensein des erm-Gens ist eine Kreuzresistenz aller Makrolide zu erwarten. Beim Vorhandensein des mef-Gens betrifft die Kreuzresistenz nur 14- und 15-gliedrige Makrolide, jedoch nicht 16-gliedrige wie Josamycin. In den letzten
Jahren wurden die 2 Resistenzmuster in unterschiedlichem Ausmaß
bei ß-hämolysierenden Streptokokken der Gruppen A, B Viele dieser Resistenz-Gene zeigen bei Streptokokken große Ähnlichkeiten, die auf mögliche Rekombinationen zwischen den Spezies hindeuten. So zeigt z.B. das mef-Gen der Pneumokokken nur einen Basen-Paar-Unterschied zum mef-Gen von S. mitis. Ziele Die Erythromycin-resistenten Stämme wurden weiter hinsichtlich ihrer Phänotypen und genetischen Marker der Makrolid-Resistenz untersucht. |
|
Die Bakterien wurden in unserem Labor in den letzten 8 Jahren aus Blutkulturen isoliert und mit dem System API 20 STREP (bioMérieux) identifiziert. Es waren 40 S. mitis, 25 S. sanguis, 11 S. salivarius, 5 S. milleri und 25 Stämme anderer Spezies. Für die Resistenztestung wurde die Agardilutions-Methode nach NCCLS angewendet. Als Nährboden wurde Mueller-Hinton-Agar mit 5% Schafblut-Zusatz und ansteigender Konzentration der gewählten Antibiotika (Clindamycin, Erythromycin, Clarithromycin, Roxithromycin, Azithromycin, Josamycin) hergestellt. Eine 1/10-Verdünnung einer Suspension, entsprechend einer Trübung von 0,5 McFarland, wurde mit einem "Multipoint Inokulator" auf die Platten aufgebracht. Die Platten wurden 20-24 Stunden bei 37°C aerob bebrütet. Für die Interpretation wurden die laut NCCLS [6] empfohlenen Grenzwertkonzentrationen angewandt und für Josamycin und Roxithromycin die von dem Comité de l' Antibiogramme de la Société Française de Microbiologie empfohlenen Grenzwerte (Tabelle 1) [7].
Der MLSB-
oder M-Phänotyp wurde mit einem Doppel-Blättchen-Test
untersucht. Erythromycin (15 µg)- und Clindamycin (2 µg)-Testblättchen
In aller Kürze: Für die Nukleinsäure-Extraktion wurde 1 ml einer über Nacht bebrüteten Bakteriensuspension 5 min bei 13.000 rpm zentrifugiert und das Pellet in 75 µl TES Puffer (20 mM TRIS pH 8, 10 mM EDTA pH8, 25% Saccharose) gelöst. Nach Zugabe von 25 µl (80 mg/ml) Lysozym wurde 1 Stunde bei 37°C inkubiert. Anschließend wurde eine Extraktion mit Säulchen der Firma QIAgen durchgeführt. Die PCR zum Nachweis der mef-, erm- und ermTR-Gene erfolgte mit der von Sutcliffe et al. [8] beschriebenen Methode. Der Restriktionsverdau zur Bestätigung der Amplifikation des ermTR-Gens wurde wie von Seppälä et al. [9] beschrieben durchgeführt. |
|
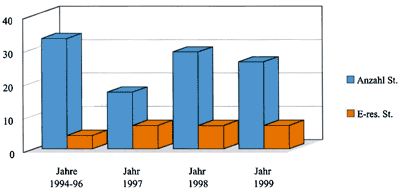
Von den 106
Stämmen vergrünender Streptokokken waren 25 (24%) Erythromycin-resistent.
Wie auch in einer anderen Untersuchung [10], die
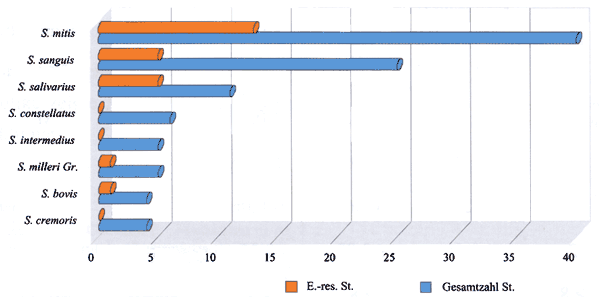
In Diagramm 2 ist die Prävalenz der Erythromycin-Resistenz bei den untersuchten vergrünenden Streptokokken nach Spezies dargestellt.
Mit dem Doppel-Blättchen-Test fanden wir 14-mal den Phänotyp M und 11-mal den Phänotyp MLSB. Mit der PCR-Methode fanden wir 12-mal das mef -, 9-mal das erm-Gen und 4-mal keines der beiden Gene. Diese letzten 4 Stämme zeigten 2-mal einen MLSB-Phänotyp und 2-mal einen M-Phänotyp (Tabelle 3 und 4).
Auch mit den ermTR-Primem (beschrieben von Seppälä et al. [9]) konnten wir das entsprechende Gen nicht nachweisen. Das Verhalten
der Erythromycin-resistenten Stämme gegenüber anderen
Makrolid-Antibiotika ist in Tabelle 4 dargestellt. Josamycin zeigt
im Aktuelle Berichte
[11] zeigen einen Anstieg der Antibiotika-Resistenz der vergrünenden
Streptokokken. Carratala et al. [12] berichteten über Die zwei wichtigeren
Makrolid-Resistenz-Mechanismen der Streptokokken wirken sich unterschiedlich
auf ihre Empfindlichkeit gegenüber Clindamycin und Josamycin
aus. Die Stämme, die ein mef-Gen besitzen, sind weiterhin
Clindamycin- und Josamycin-empfindlich. In unserer |
|
Wir fanden eine Prävalenz der Makrolid-Resistenz von 24% im untersuchten Material. Die M- und MLSB-Phänotypen waren gleich häufig repräsentiert. Außer den beschriebenen mef- und erm-Genen müssen noch andere Resistenzdeterminanten existieren. Von den 5 getesteten Makrolid-Antibiotika zeigte Josamycin die bessere Aktivität. |
| Literatur:
1. Leclercq R., Couvalin P: "Mechanism of resistance to macrolide, iincosamides, and streptogramin antibiotics by target modification." Antimicrob. Agents. Chemother. 35 (1993) 1267-1272. 2. Weisblum B.: "Erythromycin resistance by ribosome modification." Antimicrob. Agents. Chemother. 39 (1995) 577-585. 3. Seppälä H., Nissinen A., y u Q., Huovinen P.: " Three different phenotypes of erythromycin-resistant S. pyogenes in Finland." J. Antimicrob. Chemother. 32 (1993) 885-891. 4. Sutcliffe I., Tait-Kamradt A., Wondrack L.: "Streptococcus pneumoniae and Streptococcus pyogenes resistant to macrolides but sensitive to clindamycin: a common resistance pattern mediated by an efflux system." Antimicrob. Agents. Chemother. 40 (1996) 1817-1824. 5. De Azavedo I.C.S. et ai., 38th ICAAC, Sept. 24-27, 1998. 6. "Perforrnance standards for antimicrobial susceptibility testing." NCCLS, M100-S11, Vol.21, No.1 (2001) 104-105. 7. Soussy C.J., Cluzel R., Courvalin P.: "Definition and determination of in vitro antibiotic susceptibility breakpoints for bacteria in France. The Comite de I' Antibiogramme de la societe Française de Microbiologie." Eur. J. Clin. Microbiol. Infect. Dis. 13 (1994) 238-246. 8. Sutcliffe J., Grebe T., Tait-Kamradt A., Wondrack L.: "Detection of Erythromycin-Resistant Determinants by PCR." Antimicrob. Agents. Chemother. 40 (1996) 2562-2566. 9. Seppälä H., Skumik M., Soini H., Roberts M., Huovinen P.A.: "Novel Erythromycin Resistance Methylase Gene (ermTR) in Streptococcus pyogenes." Antimicrob. Agents. Chemother. 42 (1998) 257-262. 10. Jebelean C., Binder L., Haditsch M., Watschinger R., Mittermayer H.: "Antimicrobial Susceptibility of Viridans Streptococci Isolated from Blood during 1990-1998." ECCMID 1999, Berlin. 11. Renneberg J., Niemann L.L., Gutschik E. : "Antimicrobial susceptibility of 278 streptococcal blood isolates to seven antimicrobial agents." J. Antimicrob. Chemother. 39 (1997) 135-140. 12. Carratala J., Alcaide F., Femandez-Sevilla A., Corbella X., Linares J., Gudiol F.: "Bacteremia due to viridans streptococci that are highly resistant to penicillin: increase among neutropenic patients with cancer." Clin. Infect. Dis. 20 (1994) 1169-1173 13. Doern G.V, Ferraro M.J., Brueggemann A.B., Ruoff K.L.: "Emergence of high rates of antimicrobial resistance among viridans group streptococci in the United States." Antimicrob. Agents. Chemother. 40 (1996) 891-894. |
| Anschrift
der Verfasserin: Dr. Crista Jebelean Institut für Hygiene, Mikrobiologie und Tropenmedizin, Krankenhaus der Elisabethinen Linz A-4010 Linz, Fadingerstraße 1 |