| Resistenzgene in Antibiotikapräparaten – Trojanische Pferde? |
| M. Wögerbauer, H. Lagler,
H. Burgmann, W. Graninger Universitätsklinik für Innere Medizin I, Abteilung für Infektionen und Chemotherapie, AKH Wien (Leiter: Univ.-Prof. DDr. W. Graninger) |
| Schlüsselwörter:
Antibiotikaresistenz, horizontaler Gentransfer, Transformation, antimikrobielle Chemotherapie |
Zusammenfassung Multiresistente Keime verursachen zunehmend Schwierigkeiten bei der antimikrobiellen Chemotherapie. Sequenzanalysen der klinisch auffälligen Resistenzdeterminanten weisen in zahlreichen Fällen auf eine Herkunft aus den entsprechenden Antibiotikaproduzentenstämmen. Da biotechnologisch hergestellte Antibiotika mit der DNA des Produzentenstammes verunreinigt sind, sollte die Möglichkeit einer direkten Übertragung eines Resistenzgenes aus dem verabreichten Präparat zumindest diskutiert werden. Am Beispiel von Streptomycin wird der Nachweis von produzentenstammspezifischer DNA im Präparat erbracht. Mögliche Auswirkungen auf DNA-Transferprozesse in der Darmflora von Säugetieren werden skizziert. |
Key-words: Multidrug resistance, horizontal gene transfer, transformation, antibiotic preparations |
Summary The spread of multidrug resistant bacteria is severly hampering antimicrobial chemotherapy today. The basis for the rapid dissemination of resistance determinants is the ability of microorganisms to exchange genetic information readily via horizontal gene transfer. However, the origin of resistance genes, which protect clinical pathogens against antimicrobial agents, is not always clear. In some cases sequence analyses show a high degree of homology with the resistance determinants active in the producer strains of the respective antibiotic. Antibiotic preparations of microbial origin are contaminated with producer strain specific DNA. Thus, antibiotic preparations themselves may serve as a vector for the dissemination of resistance genes. With streptomycin the presence of the according producer strain specific rDNA sequences could be demonstrated. Implications on DNA transfer processes in the mammalian gastrointestinal tract are discussed. |
Einleitung Das Auftauchen multiresistenter Bakterienstämme verursacht zunehmend schwerwiegende Probleme in der antibakteriellen Chemotherapie. Motor der raschen Ausbreitung von Resistenzgenen ist die Fähigkeit von Bakterien, effizient über Speziesgrenzen hinweg genetische Information an Nachbarzellen weiterzugeben [6]. Der Ursprung klinisch auffällig werdender Resistenzdeterminanten ist häufig nur mit großem molekularbiologischen Aufwand unter Zuhilfenahme epidemiologischer Daten rückverfolgbar. Indizien sprechen jedoch zunehmend dafür, dass zumindest in manchen Fällen die neu akquirierte Resistenzdeterminante von jenen Mikroorganismen herrührt, die ursprünglich bei der biotechnologischen Produktion des Präparats eingesetzt worden sind [3, 10]. Wahrscheinlichstes Ausbreitungsszenario ist folgendes: Über einen kaskadenartigen Prozess, der zahlreiche Zwischenwirte und verschiedenartige Gentransferprozesse wie Transformation, Konjugation und Transduktion umfasst, landet die Resistenzgendeterminate in einem pathogenen Keim und interferiert dort mit der durchgeführten Chemotherapie [3]. Ist man lange Zeit davon ausgegangen, dass in ihrem natürlichen Habitat (Darm, Boden, Haut) lebende Bakterien nur selten freie DNA aufnehmen, so hat sich in den letzten Jahren das Bild völlig geändert [5]. Man muss davon ausgehen, dass nahezu jedes Bakterium unter gewissen Umweltbedingungen dazu imstande ist, freie DNA in den eigenen genetischen Apparat zu inkorporieren [4]. Diese „genetische Kompetenz“ – die Fähigkeit, DNA aus der Umgebung aufzunehmen – ist eine bei den meisten Bakterien durch Änderung des pH-Werts und Ionenmillieus, Temperaturschwankungen oder Hungerstress physiologisch induzierbare Eigenschaft [4]. Sie stellt eine Variante im prokaryotischen Repertoire dar, mit deren Hilfe Bakterienpopulationen rasch auf sich verändernde Umweltbedingungen reagieren. Der MagenDarm-Trakt von Säugetieren bietet für die Induktion kompetenter Bakterienstämme optimale Ausgangsvoraussetzungen, da hier zahlreiche der oben angeführten Umweltbedingungen anzutreffen sind. Die Applikation von Antibiotika setzt per definitionem die betroffene Bakterienpopulation unter extremen physiologischen Stress. In derartigen Situationen aktivieren Bakterien Notsysteme, die der Zelle ermöglichen könnten, dem Selektionsdruck zu widerstehen. Während einer Antibiotikatherapie werden jedoch oft nicht die notwendigen bakteriziden Konzentrationen erreicht, die zur Abtötung erforderlich wären. In niedriger Dosierung wirken Antibiotika auf Bakterien jedoch wie „Aphrodisiaka“ – Botenstoffe, die der Zelle mitteilen, sich auf den Austausch von genetischer Information vorzubereiten [3]. Mit Resistenzgenen verseuchte Antibiotika kombinieren zwei Faktoren, die die Grundlage für einen Circulus vitiosus bilden: 1. Der durch das Antibiotikum gesetzte Selektionsdruck führt per se zur Auslese resistenter Bakterienzellen, wobei unter Umständen auch Gentransfermechanismen in der betroffenen Population aktiviert werden. 2. Gleichzeitig wird mit dem kontaminierten Präparat bei oraler Applikation das entsprechende Resistenzgen in Form von freier DNA der Darmflora von Säugetieren angeboten. Welche Rolle mit produzentenstammspezifischer DNA kontaminierte Antibiotikapräparate als „Vektor“ für Resistenzgene bei deren Verbreitung tatsächlich spielen, wird kontrovers diskutiert. Wir konnten anhand von Streptomycin beispielhaft zeigen, dass im kommerziell erhältlichen Präparat genomische DNA des Produzenten Streptomyces griseus vorhanden ist. |
|
Um eventuell laborinterne DNA-Kontaminationen der untersuchten Antibiotika ausschließen zu können, wurde die DNA-Isolierung aus den Präparaten ausschließlich mit originalverpacktem und speziell für sensitive PCR-Anwendungen erhältlichem Einwegmaterial räumlich von der Baterienanzucht getrennt durchgeführt. Die Anzüchtung des Produzentenstammes (S. griseus) und dessen DNA-Extraktion erfolgte nach der Aufreinigung der Antibiotikapräparate. Der Nachweis von produzentenstammspezifischer DNA wurde mittels speziell adaptierter PCR-Protokolle durchgeführt. Primerpaare wurden für den nicht-konservierten Bereich der 16S ribosomalen DNA aus S. griseus konstruiert. Die Speziespezifität der eingesetzten Primer wurde mittels BLAST- Alignment-Studien theoretisch und durch Überprüfung an gattungsfremder DNA (z. B. E. coli) abgesichert. Kreuzkontaminationen bei den PCR-Ansätzen sind durch striktes Befolgen eines Sicherheitsprotokolls auszuschließen. |
|
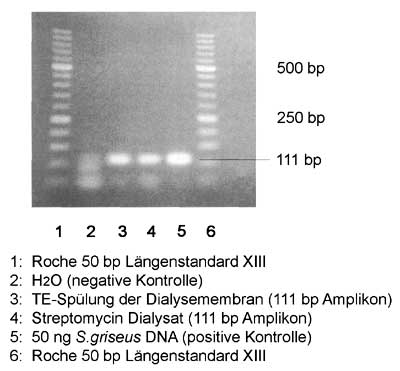
Ribosomale DNA aus Streptomyces griseus ist in Streptomycin-Präparaten nachweisbar (Abb. 1). Zu diesem Zweck wurde ein 111 bp langes DNA-Fragment als Target ausgewählt und amplifiziert. Die Detektion genomischer Produzentenstamm-DNA ist abhängig von der Fragmentlänge. Kurze DNA-Stücke werden mit einer besseren Effizienz amplifiziert als längere (Ergebnisse nicht gezeigt). Da unter Umständen beträchtliche DNA-Mengen an der verwendeten Dialysemembran haften bleiben, wurde auch eine Pufferspülung der eingesetzten Membran getestet (Abb. 1, Reihe 3). Die Nachweisführung mittels PCR ist notwendig, da Antibiotika aufgrund von (pflanzlichen) Zusatzstoffen, die bei der Präparateherstellung benötigt werden, häufig DNA enthalten. Ein DNA-Nachweis z.B. mit Hilfe von DNA-doppelstrangbindenden Fluoreszenzfarbstoffen im Präparat gibt noch keine Auskunft über die Art und Herkunft der detektierten DNA.
|
|
Relevanz der Beobachtung Um die Übertragungswahrscheinlichkeit eines Resistenzgens, das als Kontamination in einem oral verabreichten Antibiotikapräparat vorliegt, auf menschliche Darmbakterien abschätzen zu können, müssen folgende Punkte geklärt werden: 1. Wie hoch ist die Kopienzahl der gesuchten Resistenzdeterminante im Präparat (Quantität)? 2. Ist das Resistenzgen physisch intakt, womöglich mit den entsprechenden prokaryotischen Regulationselementen (Promotor, Ribosomenbindungsstelle, Terminator, Replikationsursprung) im Präparat nachzuweisen (Qualität)? 3. Übersteht doppelsträngige DNA die Magen-Darm-Passage bei Säugetieren unbeschadet (Protektion)? 4. Besteht die Möglichkeit, dass oral verabreichte DNA im Gastrointestinaltrakt auf DNA-aufnahmefähige Mikroorganismen trifft (bakterielle Kompetenz)? Wie gezeigt werden konnte, sind mikrobiologisch hergestellte Antibiotikapräparate zum Teil beträchtlich mit doppelsträngiger Desoxyribonukleinsäure verunreinigt [10]. Die fluorometrisch bestimmten DNA-Mengen pro Gramm Antibiotikum würden durchaus ausreichen, um lokal im Darm Konzentrationen zu generieren, die einen effektiven Gentransfer auf bakterielle Wirtszellen gestatten. Die im Präparat detektierte Gesamt-DNA besteht jedoch nur zu einem Bruchteil aus genomischer DNA des Produzentenstammes und liegt zudem stark fragmentiert vor. Ribosomale Produzentenstamm-DNA ist im gewählten PCR-Setting relativ leicht zu detektieren, trotzdem ist eine Abnahme der Effizienz mit zunehmender Fragmentlänge zu beobachten. Der Resistenzgenfragmentnachweis ist nur mit speziell adaptierten PCR-Methoden zu erbringen. Diese Beobachtungen deuten darauf hin, dass im Präparat äußerst selten voll funktionstüchtige Resistenzgene anzutreffen sein dürften. |
|
Antibiotika in der Tierzucht Die Anwendung von Antibiotika-Präparaten als Wachstumspromotoren in der Tierzucht stellt ein deutlich höheres Risikopotential dar, da hier ständig über lange Zeiträume Substanzen in subtherapeutischen Konzentrationen zugeführt werden. Die Wahrscheinlichkeit, dass in diesem Szenario auch Resistenzgene aus kontaminierten Präparaten transferiert werden, ist deutlich höher als bei humantherapeutischen Anwendungen. Die Generation von Resistenzgenpools in der Landwirtschaft und Tierzucht durch den permanenten Selektionsdruck steht außer Zweifel [7]. Humanpathogene Keime haben aufgrund des offenen mikrobiellen Ökosystems Zugriff auf diesen Resistenzgenpool. Der Zugriff kann in Form einer direkten Übertragung zoonotischer Keime in das humane Habitat, aber auch indirekt durch den Transfer von einzelnen Resistenzdeterminaten in einem kaskadenartigen Prozess mit zahlreichen Zwischenwirten auf humanpathogene Keime erfolgen [3, 7]. |
| Literatur:
1. Bauer F., Hertel C., Hammes
W.P.: „Transformation of E. coli in foodstuffs.“ System. Appl. Microbiol.
22 (1999) 161-168. |
| Anschrift
des Verfassers: Univ-Prof. DDr. W. Graninger Univ.-Klinik für Innere Medizin I, Abt. für Infektionen und Chemotherapie A-1090 Wien, Währinger Gürtel 18-20 |